1번
원점을 지나는 회귀모형은 다음과 같이 정의할 수 있다.
\[y_i = β_1x_i + ϵ_i, ϵ_i ∼_{i.i.d.} N(0, σ^2), i = 1, \dots , n\]
(1)
오차제곱합을 정의하고 \(β_1\)의 최소제곱추정량 \((\hatβ_1)\)을 구하여라.
\(S=\sum_{i=1}^n \epsilon^2 = \sum_{i=1}^n (y_i-\beta_1 x_i)^2 = \sum(y_i^2 + \beta_1^2 x_i^2 -2 \beta_1 x_i y_i)\)
\(\widehat \beta_1 = argmin \sum_{i=1}^n(y_i - \beta_1 x_i)^2\)
\(\dfrac{\partial S}{\partial \beta_1}= -2 \sum_{i=1}^n x_i(y_i-\beta_1 x_i)\)
= \(\sum x_iy_i - \beta_1 \sum x_i^2 = 0\)
\(\therefore \widehat \beta_1 = \dfrac{\sum_{i=1}^n x_i y_i}{\sum_{i=1}^n x_i^2}\)
(2)
\(E(\hatβ_1)\)을 구하여라.
\(a_i = \dfrac{x_i}{\sum_{i=1}^n x_i^2}\)라고 놓자.
즉, \(\widehat \beta_1 = \sum_{i=1}^n a_i y_i\)
\(E(\widehat \beta_1)= E(\sum_{i=1}^n a_i y_i) = \sum_{i=1}^n a_i E(y_i)=\sum_{i=1}^n a_i E(\beta_1 x_i + \epsilon_i)=\sum_{i=1}^n a_i \beta_1 x_i = \dfrac{\sum_{i=1}^n x_i^2}{\sum_{i=1}^n x_i^2} \beta_1 = \beta_1\)
\(E(\hatβ_1)=\beta_1\)이므로 불편추정량
(3)
\(Var(\hatβ_1)\)을 구하여라.
\(Var(\widehat \beta_1)= Var(\sum_{i=1}^n a_i y_i) = \sum_{i=1}^n a_i^2 Var(y_i)=\dfrac{\sigma^2}{\sum_{i=1}^n x_i^2}\)
\(\because Var(y_i) = \sigma^2\)
(4)
제곱합에 대한 분산분석표를 작성하여라.
| 회귀 |
\(SSR\) |
1 |
\(MSR=\dfrac{SSR}{1}\) |
\(\dfrac{MSR}{MSE}\) |
\(P(F \geq F_0)\) |
| 잔차 |
\(SSE\) |
\(n-1\) |
\(MSE=\dfrac{SSE}{n-1}\) |
|
|
| 계 |
\(SST\) |
\(n\) |
|
|
|
\(SSR=\sum_{i=1}^n (\widehat y_i)^2 = \sum (\widehat \beta_1 x_i)^2 = \widehat \beta_1^2 \sum x_i^2\)
\(SSE=\sum_{i=1}^n(y_i - \widehat y)^2 = \sum(y_i - \widehat \beta_1 x_i)^2 = \sum y_i^2 - SSR\)
\(SST=SSE+SSR=\sum y_i^2\)
절편이 없는 모형은 평균이 0인 느낌
\(R^2=\dfrac{\sum \widehat y_i^2}{\sum y_i^2}\)
(5)
회귀모형의 유의성 검정을 하기 위한 가설을 설정하고, 검정통계량을 제시하여라.
가설 \(H_0: \beta_1 = 0 \ vs \ H_1:\beta_1 \neq 0\)
검정통계량 \(F=\dfrac{MSR}{MSE}=\dfrac{SSR/1}{SSE/(n-1)} \sim_{H_0} F(1,n-1)\)
(6)
위의 가설에 대해, 유의수준 \(α\)에서 검정하는 방법을 기술하여라.
\(F_0 > F_\alpha(1,n-1)\)이면 귀무가설을 기각(유의함)하고 그 외는 귀무가설을 채택
혹은,
유의확률 = \(P(F>F_0) < \alpha \to H_0\)기각
유의확률 = \(P(F>F_0) > \alpha \to H_0\)기각못함
(7)
다음의 가설에 대한 검정통계량을 제시하고, 유의수준 \(α\)에서 가설 검정하는 방법을 기술하여라.
\[H_0 : β_1 = 0 \ vs \ H_1 : β_1 > 0\]
검정통계량 \(T=\dfrac{\widehat \beta_1 - 0}{\widehat{s.e}(\widehat \beta_1)} \sim_{H_0} t(n-1)\)
\(s.e(\widehat \beta_1)=\sqrt{Var(\widehat \beta_1)} = \dfrac{\sigma}{\sqrt{\sum x_i^2}}\)
\(\widehat{s.e}(\widehat \beta_1)= \sqrt{\dfrac{\hat {\sigma^2}}{\sum x_i^2}}, {\hat \sigma^2}=MSE\) $
유의확률 = \(P(T > t_0) < \alpha \to H_0\)기각
혹은
\(t_0>t_{\alpha}(n-1) \to H_0\) 기각
\(t_0<t_{\alpha}(n-1) \to H_0\) 기각 못함
2번
‘cars.csv’ 데이터를 이용하여 회귀모형을 적합하려고 한다.
이는 자동차의 속도(mph)에 따른 제동거리(ft)를 조사한 데이터이다.
다음 물음에 답하여라. (R을 이용하여 풀이)(검정에서는 유의수준 \(α = 0.05\) 사용)
── Attaching packages ─────────────────────────────────────── tidyverse 1.3.1 ──
✔ ggplot2 3.4.1 ✔ purrr 1.0.1
✔ tibble 3.2.0 ✔ dplyr 1.1.0
✔ tidyr 1.3.0 ✔ stringr 1.5.0
✔ readr 2.1.4 ✔ forcats 1.0.0
── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
ERROR: Error: 'cars' does not exist in current working directory ('/home/coco/Dropbox/coco/posts/Applied statistics').
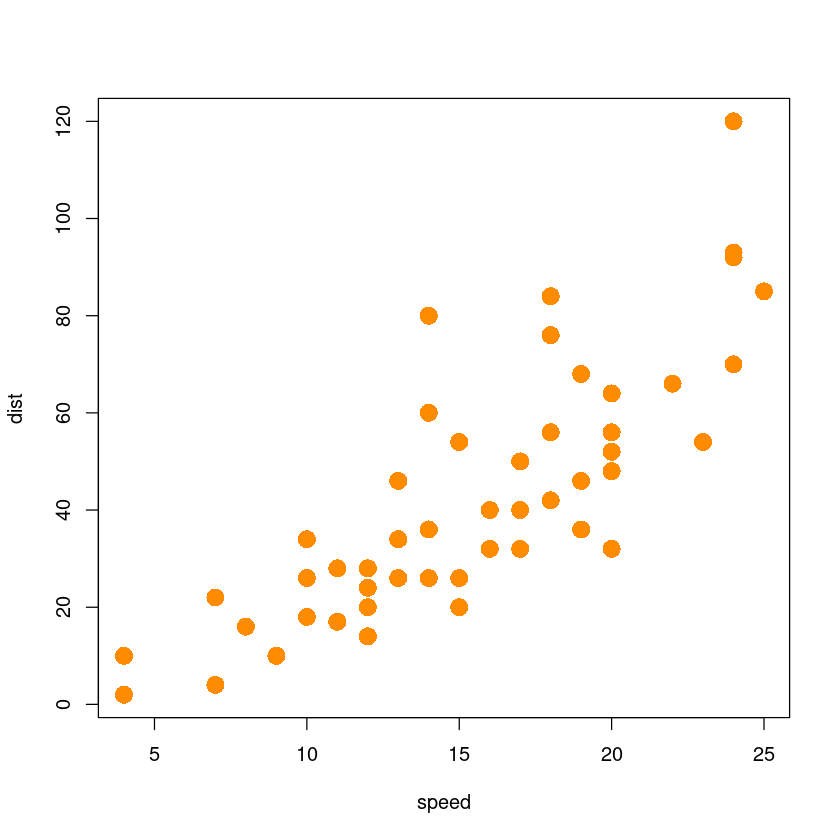
(1) 산점도
이 데이터의 산점도를 그리고 두 변수 사이의 관계를 설명하시오.
plot(dist~speed,
data=cars,
xlab="speed",
ylab="dist",
pch=16,
cex=2,
col="darkorange")
(2) 회귀직선
최소제곱법의 의한 회귀직선을 적합시키시키고, 모형 적합 결과를 설명하시오.
dt <- data.frame(
i = 1:nrow(cars),
x = cars$speed,
y = cars$dist,
x_barx = cars$speed - mean(cars$speed),
y_bary = cars$dist - mean(cars$dist))
dt
A data.frame: 50 × 5
| <int> |
<dbl> |
<dbl> |
<dbl> |
<dbl> |
| 1 |
4 |
2 |
-11.4 |
-40.98 |
| 2 |
4 |
10 |
-11.4 |
-32.98 |
| 3 |
7 |
4 |
-8.4 |
-38.98 |
| 4 |
7 |
22 |
-8.4 |
-20.98 |
| 5 |
8 |
16 |
-7.4 |
-26.98 |
| 6 |
9 |
10 |
-6.4 |
-32.98 |
| 7 |
10 |
18 |
-5.4 |
-24.98 |
| 8 |
10 |
26 |
-5.4 |
-16.98 |
| 9 |
10 |
34 |
-5.4 |
-8.98 |
| 10 |
11 |
17 |
-4.4 |
-25.98 |
| 11 |
11 |
28 |
-4.4 |
-14.98 |
| 12 |
12 |
14 |
-3.4 |
-28.98 |
| 13 |
12 |
20 |
-3.4 |
-22.98 |
| 14 |
12 |
24 |
-3.4 |
-18.98 |
| 15 |
12 |
28 |
-3.4 |
-14.98 |
| 16 |
13 |
26 |
-2.4 |
-16.98 |
| 17 |
13 |
34 |
-2.4 |
-8.98 |
| 18 |
13 |
34 |
-2.4 |
-8.98 |
| 19 |
13 |
46 |
-2.4 |
3.02 |
| 20 |
14 |
26 |
-1.4 |
-16.98 |
| 21 |
14 |
36 |
-1.4 |
-6.98 |
| 22 |
14 |
60 |
-1.4 |
17.02 |
| 23 |
14 |
80 |
-1.4 |
37.02 |
| 24 |
15 |
20 |
-0.4 |
-22.98 |
| 25 |
15 |
26 |
-0.4 |
-16.98 |
| 26 |
15 |
54 |
-0.4 |
11.02 |
| 27 |
16 |
32 |
0.6 |
-10.98 |
| 28 |
16 |
40 |
0.6 |
-2.98 |
| 29 |
17 |
32 |
1.6 |
-10.98 |
| 30 |
17 |
40 |
1.6 |
-2.98 |
| 31 |
17 |
50 |
1.6 |
7.02 |
| 32 |
18 |
42 |
2.6 |
-0.98 |
| 33 |
18 |
56 |
2.6 |
13.02 |
| 34 |
18 |
76 |
2.6 |
33.02 |
| 35 |
18 |
84 |
2.6 |
41.02 |
| 36 |
19 |
36 |
3.6 |
-6.98 |
| 37 |
19 |
46 |
3.6 |
3.02 |
| 38 |
19 |
68 |
3.6 |
25.02 |
| 39 |
20 |
32 |
4.6 |
-10.98 |
| 40 |
20 |
48 |
4.6 |
5.02 |
| 41 |
20 |
52 |
4.6 |
9.02 |
| 42 |
20 |
56 |
4.6 |
13.02 |
| 43 |
20 |
64 |
4.6 |
21.02 |
| 44 |
22 |
66 |
6.6 |
23.02 |
| 45 |
23 |
54 |
7.6 |
11.02 |
| 46 |
24 |
70 |
8.6 |
27.02 |
| 47 |
24 |
92 |
8.6 |
49.02 |
| 48 |
24 |
93 |
8.6 |
50.02 |
| 49 |
24 |
120 |
8.6 |
77.02 |
| 50 |
25 |
85 |
9.6 |
42.02 |
dt$x_barx2 <- dt$x_barx^2
dt$y_bary2 <- dt$y_bary^2
dt$x_barxy_bary <-dt$x_barx * dt$y_bary
dt
A data.frame: 50 × 8
| <int> |
<dbl> |
<dbl> |
<dbl> |
<dbl> |
<dbl> |
<dbl> |
<dbl> |
| 1 |
4 |
2 |
-11.4 |
-40.98 |
129.96 |
1679.3604 |
467.172 |
| 2 |
4 |
10 |
-11.4 |
-32.98 |
129.96 |
1087.6804 |
375.972 |
| 3 |
7 |
4 |
-8.4 |
-38.98 |
70.56 |
1519.4404 |
327.432 |
| 4 |
7 |
22 |
-8.4 |
-20.98 |
70.56 |
440.1604 |
176.232 |
| 5 |
8 |
16 |
-7.4 |
-26.98 |
54.76 |
727.9204 |
199.652 |
| 6 |
9 |
10 |
-6.4 |
-32.98 |
40.96 |
1087.6804 |
211.072 |
| 7 |
10 |
18 |
-5.4 |
-24.98 |
29.16 |
624.0004 |
134.892 |
| 8 |
10 |
26 |
-5.4 |
-16.98 |
29.16 |
288.3204 |
91.692 |
| 9 |
10 |
34 |
-5.4 |
-8.98 |
29.16 |
80.6404 |
48.492 |
| 10 |
11 |
17 |
-4.4 |
-25.98 |
19.36 |
674.9604 |
114.312 |
| 11 |
11 |
28 |
-4.4 |
-14.98 |
19.36 |
224.4004 |
65.912 |
| 12 |
12 |
14 |
-3.4 |
-28.98 |
11.56 |
839.8404 |
98.532 |
| 13 |
12 |
20 |
-3.4 |
-22.98 |
11.56 |
528.0804 |
78.132 |
| 14 |
12 |
24 |
-3.4 |
-18.98 |
11.56 |
360.2404 |
64.532 |
| 15 |
12 |
28 |
-3.4 |
-14.98 |
11.56 |
224.4004 |
50.932 |
| 16 |
13 |
26 |
-2.4 |
-16.98 |
5.76 |
288.3204 |
40.752 |
| 17 |
13 |
34 |
-2.4 |
-8.98 |
5.76 |
80.6404 |
21.552 |
| 18 |
13 |
34 |
-2.4 |
-8.98 |
5.76 |
80.6404 |
21.552 |
| 19 |
13 |
46 |
-2.4 |
3.02 |
5.76 |
9.1204 |
-7.248 |
| 20 |
14 |
26 |
-1.4 |
-16.98 |
1.96 |
288.3204 |
23.772 |
| 21 |
14 |
36 |
-1.4 |
-6.98 |
1.96 |
48.7204 |
9.772 |
| 22 |
14 |
60 |
-1.4 |
17.02 |
1.96 |
289.6804 |
-23.828 |
| 23 |
14 |
80 |
-1.4 |
37.02 |
1.96 |
1370.4804 |
-51.828 |
| 24 |
15 |
20 |
-0.4 |
-22.98 |
0.16 |
528.0804 |
9.192 |
| 25 |
15 |
26 |
-0.4 |
-16.98 |
0.16 |
288.3204 |
6.792 |
| 26 |
15 |
54 |
-0.4 |
11.02 |
0.16 |
121.4404 |
-4.408 |
| 27 |
16 |
32 |
0.6 |
-10.98 |
0.36 |
120.5604 |
-6.588 |
| 28 |
16 |
40 |
0.6 |
-2.98 |
0.36 |
8.8804 |
-1.788 |
| 29 |
17 |
32 |
1.6 |
-10.98 |
2.56 |
120.5604 |
-17.568 |
| 30 |
17 |
40 |
1.6 |
-2.98 |
2.56 |
8.8804 |
-4.768 |
| 31 |
17 |
50 |
1.6 |
7.02 |
2.56 |
49.2804 |
11.232 |
| 32 |
18 |
42 |
2.6 |
-0.98 |
6.76 |
0.9604 |
-2.548 |
| 33 |
18 |
56 |
2.6 |
13.02 |
6.76 |
169.5204 |
33.852 |
| 34 |
18 |
76 |
2.6 |
33.02 |
6.76 |
1090.3204 |
85.852 |
| 35 |
18 |
84 |
2.6 |
41.02 |
6.76 |
1682.6404 |
106.652 |
| 36 |
19 |
36 |
3.6 |
-6.98 |
12.96 |
48.7204 |
-25.128 |
| 37 |
19 |
46 |
3.6 |
3.02 |
12.96 |
9.1204 |
10.872 |
| 38 |
19 |
68 |
3.6 |
25.02 |
12.96 |
626.0004 |
90.072 |
| 39 |
20 |
32 |
4.6 |
-10.98 |
21.16 |
120.5604 |
-50.508 |
| 40 |
20 |
48 |
4.6 |
5.02 |
21.16 |
25.2004 |
23.092 |
| 41 |
20 |
52 |
4.6 |
9.02 |
21.16 |
81.3604 |
41.492 |
| 42 |
20 |
56 |
4.6 |
13.02 |
21.16 |
169.5204 |
59.892 |
| 43 |
20 |
64 |
4.6 |
21.02 |
21.16 |
441.8404 |
96.692 |
| 44 |
22 |
66 |
6.6 |
23.02 |
43.56 |
529.9204 |
151.932 |
| 45 |
23 |
54 |
7.6 |
11.02 |
57.76 |
121.4404 |
83.752 |
| 46 |
24 |
70 |
8.6 |
27.02 |
73.96 |
730.0804 |
232.372 |
| 47 |
24 |
92 |
8.6 |
49.02 |
73.96 |
2402.9604 |
421.572 |
| 48 |
24 |
93 |
8.6 |
50.02 |
73.96 |
2502.0004 |
430.172 |
| 49 |
24 |
120 |
8.6 |
77.02 |
73.96 |
5932.0804 |
662.372 |
| 50 |
25 |
85 |
9.6 |
42.02 |
92.16 |
1765.6804 |
403.392 |
- i
- 1275
- x
- 770
- y
- 2149
- x_barx
- -1.77635683940025e-14
- y_bary
- 1.63424829224823e-13
- x_barx2
- 1370
- y_bary2
- 32538.98
- x_barxy_bary
- 5387.4
\(\beta_1 = \dfrac{S_{xy}}{S_{xx}}\)
\(\beta_0 = \bar y - \beta_1 \bar x\)
beta1 <- as.numeric(colSums(dt)[8]/colSums(dt)[6])
beta0 <- mean(cars$dist) - beta1 * mean(cars$speed)
beta1
beta0
3.93240875912409
-17.579094890511
model <- lm(dist~speed, cars)
model
Call:
lm(formula = dist ~ speed, data = cars)
Coefficients:
(Intercept) speed
-17.579 3.932
Call:
lm(formula = dist ~ speed, data = cars)
Residuals:
Min 1Q Median 3Q Max
-29.069 -9.525 -2.272 9.215 43.201
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -17.5791 6.7584 -2.601 0.0123 *
speed 3.9324 0.4155 9.464 1.49e-12 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 15.38 on 48 degrees of freedom
Multiple R-squared: 0.6511, Adjusted R-squared: 0.6438
F-statistic: 89.57 on 1 and 48 DF, p-value: 1.49e-12
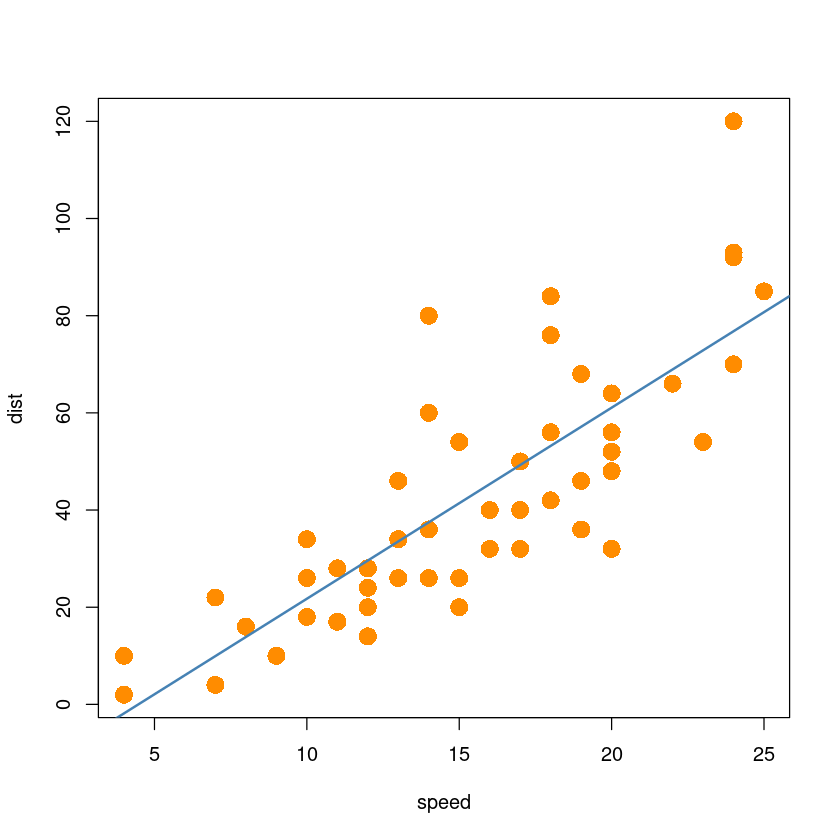
(3) 산점도 위에 회귀직선
데이터의 산점도를 그리고 추정한 회귀직선을 (1)에서 그린 산점도 위에 그리시오.
plot(dist~speed,
data=cars,
xlab="speed",
ylab="dist",
pch=16,
cex=2,
col="darkorange")
abline(model, col='steelblue', lwd=2)
(4) 분산분석
분산분석표를 작성하고 회귀직선의 유의 여부를 검정하시오.
A anova: 2 × 5
|
<int> |
<dbl> |
<dbl> |
<dbl> |
<dbl> |
| speed |
1 |
21185.46 |
21185.4589 |
89.56711 |
1.489836e-12 |
| Residuals |
48 |
11353.52 |
236.5317 |
NA |
NA |
- 가정: \(H_0: \beta_1 = 0\) vs \(H_1: \beta_1 \neq 0\)
- \(F_0 > F_{0.05}(0.95,1,48) = 4.04\) 이므로 귀무가설을 기각할 수 잇다. 즉 회귀직선이 유의하다.
(5) 결정계수, 상관계수
결정계수와 상관계수를 구하고 이 둘의 관계를 설명하시오.
\(\mathbb{R^2} = \dfrac{SSR}{SST} = \dfrac{21185.46}{32538.98} = 0.651079413060889\)
- 결정계수 직접계산
SST = sum((dt$y- mean(dt$y))^2)
SSR = sum(((-17.57909 + 3.932409*dt$x)-mean(dt$y))^2)
32538.98
21185.4615442987
- 결정계수 코드
\(r_{xy} = \dfrac{S_{xy}}{\sqrt{S{(xx)}S{(yy)}}}\)
Sxy <- sum((dt$x - mean(dt$x))*(dt$y - mean(dt$y)))
Sxx <- sum((dt$x - mean(dt$x))^2)
Syy <- sum((dt$y - mean(dt$y))^2)
단순선형회귀모형에서는 표본상관계수와 결정계수가 같다.
\(\mathbb{R^2} = r_{xy}^2\)
(6) 개별 회귀계수 유의성검정
\(β_0, β_1\)에 대한 개별 회귀계수의 유의성검정을 수행하시오.
가설 \(H_0: \beta_1 = 0\) vs \(H_1: not H_0\)
- 직접구현
tvalue1 = beta1/(sqrt((MSE/sum((dt$x-mean(dt$x))^2))))
tvalue1
9.46399107202371
tvalue0 = beta0/(sqrt((MSE*((1/48)+((mean(dt$x))^2/sum((dt$x-mean(dt$x))^2))))))
tvalue0
-2.59546417223485
- 코드구현
A matrix: 2 × 4 of type dbl
| (Intercept) |
-17.579095 |
6.7584402 |
-2.601058 |
1.231882e-02 |
| speed |
3.932409 |
0.4155128 |
9.463990 |
1.489836e-12 |
- 결과
\(\beta_0\)에 대한 \(t-vlaue\)값이 \(-2.601058\)
\(\beta_1\)에 대한 \(t-vlaue\)값이 \(9.463990\)
\(\beta_0, \beta_1\)의 t-value는 유의수준 \(\alpha=0.05\)에서의 \(tvalue=-2.011\)보다 크기 때문에 유의하다. 즉 귀무가설을 기각한다. \(\beta_0, \beta_1\)은 모두 0이 아니다.
(7) 개별 회귀계수 신뢰구간
\(β_0, β_1\)에 대한 90% 신뢰구간을 구하시오.
\[\widehat \beta_0 \pm t_{\alpha/2}(n-2) \widehat \sigma \sqrt{\dfrac{1}{n}+\dfrac{\bar x^2}{S_{xx}}}\]
\[\widehat \beta_1 \pm t_{\alpha/2}(n-2) \dfrac{\widehat \sigma}{\sqrt{S_{xx}}}\]
confint(model, level=0.9)
A matrix: 2 × 2 of type dbl
| (Intercept) |
-28.914514 |
-6.243676 |
| speed |
3.235501 |
4.629317 |
coef(model) + qt(0.95, 48) * summary(model)$coef[,2]
- (Intercept)
- -6.24367551036937
- speed
- 4.62931684193222
coef(model) - qt(0.95, 48) * summary(model)$coef[,2]
- (Intercept)
- -28.9145142706524
- speed
- 3.23550067631595
(8) 평균반응, 신뢰구간
속도가 18.5mph 인 차량의 평균 제동거리를 예측하고, 95% 신뢰구간을 구하시오.
개별 speed = 18,5
new_speed <- data.frame(speed=18.5)
- 코드
model$coefficients[1] + model$coefficients[2]*18.5
(Intercept): 55.1704671532847
predict(model,
newdata = new_speed,
interval = c("confidence"), #구간추정
level = 0.95) ##평균반응
A matrix: 1 × 3 of type dbl
| 1 |
55.17047 |
50.08797 |
60.25296 |
- 직접계산
- 평균반응 추정량
\[\widehat \mu_0 = \widehat \beta_0 + \widehat \beta_1 x_0\]
mu0 = beta0+beta1*18.5
mu0
55.1704671532847
\[Var(\widehat\mu_0))=\sigma^2(\dfrac{1}{n} + \dfrac{(x_0 - \bar x)^2}{S_{xx}})\]
varmu=((MSE*((1/48)+((18.5-mean(dt$x))^2/sum((dt$x-mean(dt$x))^2)))))
semu=sqrt(varmu)
(9) 개별반응, 신뢰구간
속도가 18.5mph 인 차량의 개별 제동거리를 예측하고, 95% 신뢰구간을 구하시오.
- 코드
predict(model, newdata = new_speed,
interval = c("prediction"),
level = 0.95) ## 개별 y
A matrix: 1 × 3 of type dbl
| 1 |
55.17047 |
23.83284 |
86.5081 |
- 직접계산
vary0=MSE*(1+(1/48)+(18.5-mean(dt$x))^2/sum((dt$x-mean(dt$x))^2))
vary0
243.118551337083
mu0+qt(0.975,48)*sqrt(vary0)
86.5208057329061
mu0-qt(0.975,48)*sqrt(vary0)
23.8201285736632
(10) 원점 지나는 회귀직선
원점을 지나는 회귀직선을 구하시오.
- 코드
model2 <- lm(dist ~ 0 + speed, cars)
summary(model2)
Call:
lm(formula = dist ~ 0 + speed, data = cars)
Residuals:
Min 1Q Median 3Q Max
-26.183 -12.637 -5.455 4.590 50.181
Coefficients:
Estimate Std. Error t value Pr(>|t|)
speed 2.9091 0.1414 20.58 <2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 16.26 on 49 degrees of freedom
Multiple R-squared: 0.8963, Adjusted R-squared: 0.8942
F-statistic: 423.5 on 1 and 49 DF, p-value: < 2.2e-16
\(\widehat {dist} = 2.9091 \widehat {speed}\)
- 직접계산
beta1_0 <- sum(dt$x * dt$y)/sum((dt$x^2))
beta1_0
2.9091321439371
(11) 원점 지나는 회귀계수 신뢰구간
위 회귀직선에서 회귀계수(기울기)의 90% 신뢰구간을 구하시오.
-코드
confint(model2, level=0.9)
A matrix: 1 × 2 of type dbl
| speed |
2.67212 |
3.146144 |
- 직접계산
SSR_0 = sum(((2.909132*dt$x))^2 )
SSE_0 = sum((dt$y - 2.909132*dt$x)^2)
beta1_0 + qt(0.95,49) * sigma_0/sqrt(1370)
3.64560479353767
beta1_0 - qt(0.95,49) * sigma_0/sqrt(1370)
2.17265949433653
(12) 원점 지난 회귀직선 분산분석
원점을 지나는 회귀직선에 대한 분산분석표를 작성하고, 회귀직선의 유의 여부를 검정하시오.
A anova: 2 × 5
|
<int> |
<dbl> |
<dbl> |
<dbl> |
<dbl> |
| speed |
1 |
111949.22 |
111949.2232 |
423.4682 |
9.227817e-26 |
| Residuals |
49 |
12953.78 |
264.3628 |
NA |
NA |
- 가정: \(H_0: \beta_1 = 0\) vs \(H_1: \beta_1 \neq 0\)
- \(F_0 > F_{0.05}(0.95,1,49) = 4.04\) 이므로 귀무가설을 기각할 수 잇다. 즉 회귀직선이 유의하다.
(13) 원점 지나는 회귀직선 결정계수
원점을 지나는 회귀직선의 결정계수를 구하시오.
- 코드
summary(model2)$r.squared
0.896289305805206
- 직접계산
(14) 회귀직선 결과 비교
원점을 포함한 회귀직선과 포함하지 않은 회귀직선의 결과를 비교하여라.
summary(model)$r.squared
summary(model2)$r.squared
0.651079380758251
0.896289305805206
원점을 포함한 회귀직선의 R스퀘어값이 더 크므로 model2가 더 좋은 것 같다.
(15) 잔차 산점도
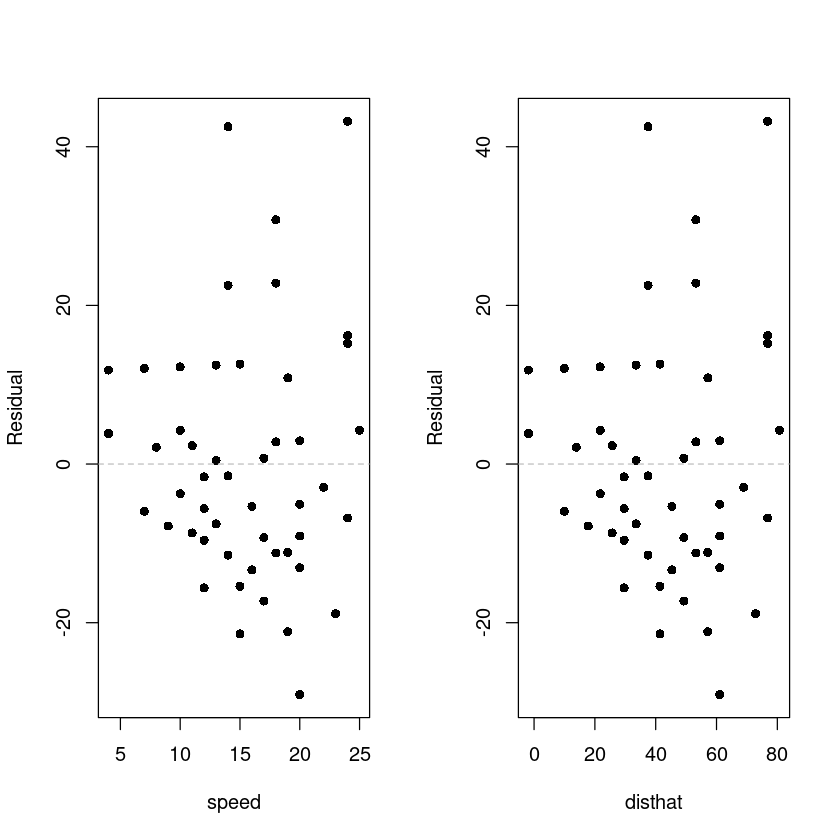
잔차에 대한 산점도를 그리고, 결과를 설명하여라.
cars$disthat <- model$fitted
cars$resid <- model$residuals
par(mfrow=c(1,2))
plot(resid ~ speed, cars, pch=16, ylab = 'Residual')
abline(h=0, lty=2, col='grey')
plot(resid ~ disthat, cars, pch=16, ylab = 'Residual')
abline(h=0, lty=2, col='grey')
(16) 잔차 등분산성 검정
잔차에 대한 등분산성 검정을 수행하시오.
가설:
studentized Breusch-Pagan test
data: model
BP = 3.2149, df = 1, p-value = 0.07297
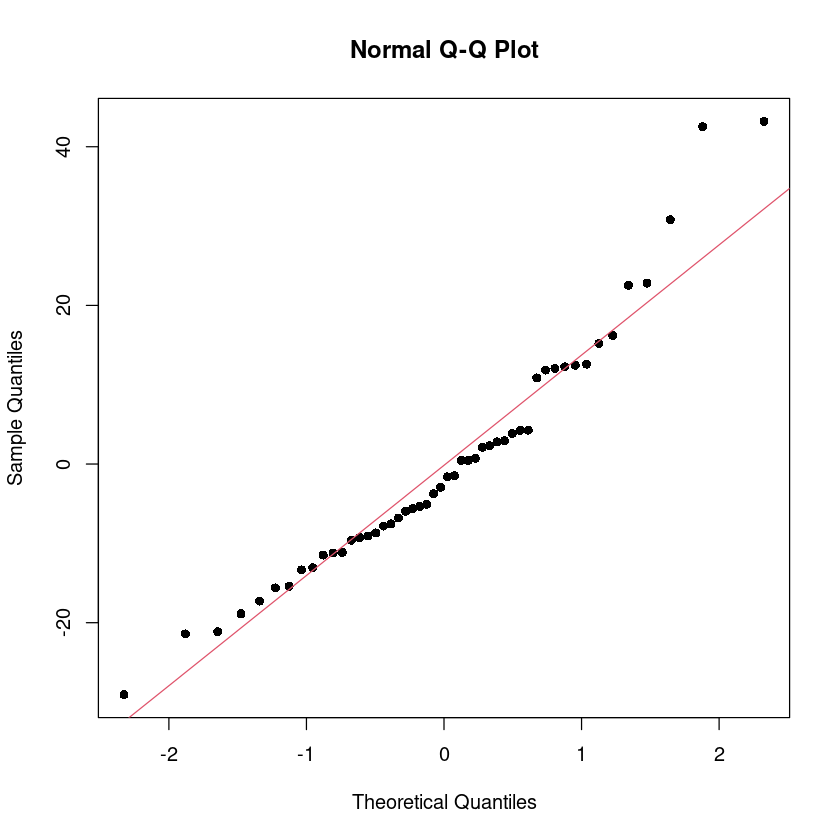
(17) 잔차 정규성 검정
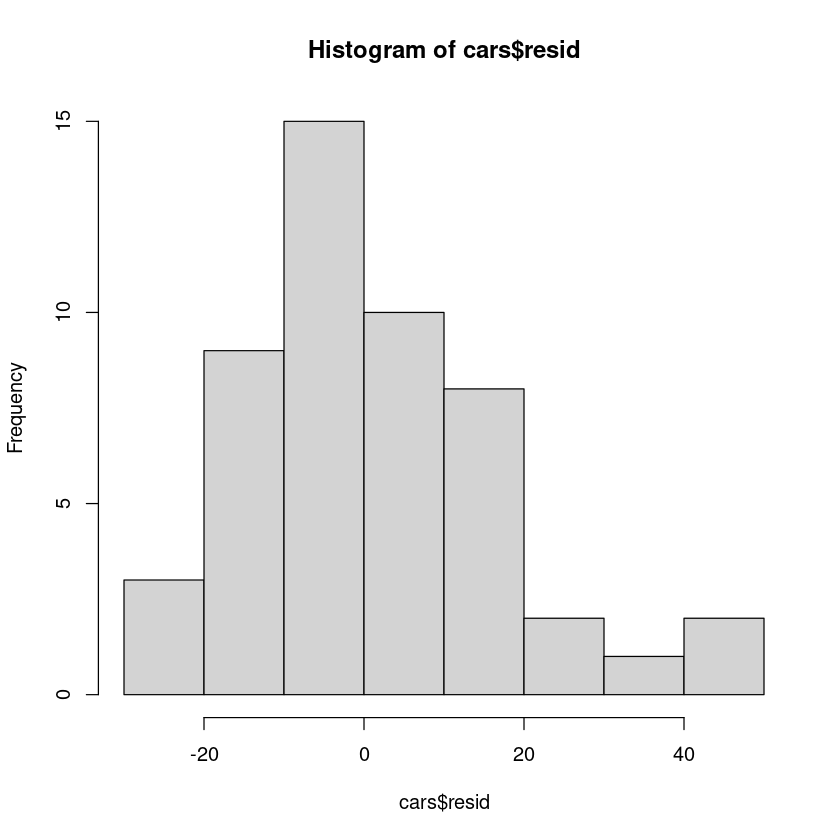
잔차에 대한 히스토그램, QQ plot을 그리고, 정규성 검정을 수행하여라.
가설:
qqnorm(cars$resid, pch=16)
qqline(cars$resid, col=2)
hist(cars$resid)
shapiro.test(resid(model))
Shapiro-Wilk normality test
data: resid(model)
W = 0.94509, p-value = 0.02152
(18) 잔차 독립성 검정
잔차에 대한 독립성 검정을 수행하시오.
dwtest(model, alternative = "two.sided") #H0 : uncorrelated vs H1 : rho != 0
Durbin-Watson test
data: model
DW = 1.6762, p-value = 0.1904
alternative hypothesis: true autocorrelation is not 0
- DW TEST의 p-value=0.1904이므로 H0를 기각할 수 없다. 채택. 즉 서로 독립이다.
dwtest(model, alternative = "greater") #H0 : uncorrelated vs H1 : rho > 0
Durbin-Watson test
data: model
DW = 1.6762, p-value = 0.09522
alternative hypothesis: true autocorrelation is greater than 0
dwtest(model, alternative = "less") #H0 : uncorrelated vs H1 : rho < 0
Durbin-Watson test
data: model
DW = 1.6762, p-value = 0.9048
alternative hypothesis: true autocorrelation is less than 0